UniFrac distance 的優缺點是什麼?
本文簡介 UniFrac 的意義與計算方式,並以其他文獻補充 Robert Edgar 對 UniFrac 的評論。
UniFrac distance 是什麼?
UniFrac 簡介
UniFrac (Unique Fraction distance) 是基於系統發生距離的 β 多樣性指標,群集間各族群的系統發生距離愈遠,表示兩群集的差異愈大, β 多樣性也愈高。UniFrac 得用以計算不同環境的微生物相差異,或是觀察干擾前後的轉變。
由於度量 UniFrac 的單位是標的基因序列(例如:16S rRNA 變異區),所以能迴避微生物模糊的物種概念。此外,序列差異與微生物的表徵有關。因此,相較於對不同序列等量齊觀的分類基礎指標 (taxon-based metrics),採計序列歧異度的 UniFrac 能充分利用每個鹼基的資訊。
Unweighted UniFrac
依據採計族群規模與否,UniFrac distance 可分為 unweighted UniFrac(不採計)和 weighted UniFrac(採計)。unweighted UniFrac 可直觀地理解為,系統發生樹被單一群集獨佔的比率。獨占枝長的比率愈多,表示兩群集成員的親緣關係愈遠,群集間的差異也愈大。
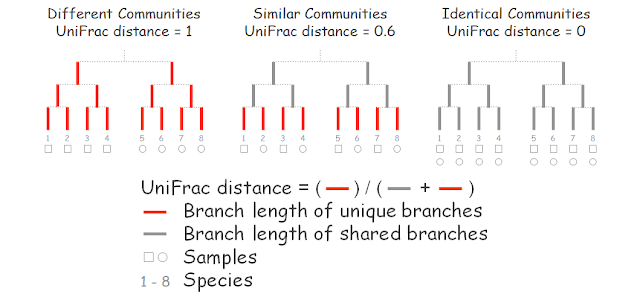
unweighted UniFrac 的計算可簡化為:(1) 標註系統發生樹上各枝所屬群集,(2) 計算獨佔枝長和總枝長的比值。
Weighted UniFrac
然而,優勢物種和稀有物種在生態學上的含義不同,如果某共享枝底下兩群集的成員比例懸殊,或許要採計族群規模以發現群集間的定量差異。因此 weighted UniFrac 依照兩群集成員的相對數量差異,量化每條枝的獨佔程度。
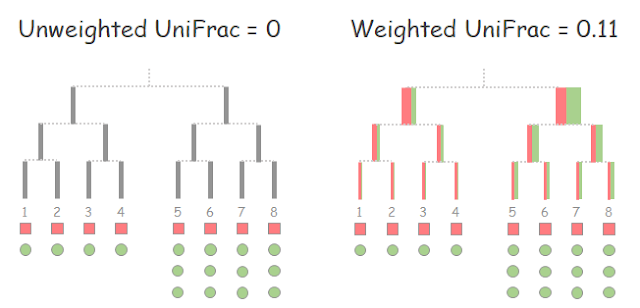
幾何圖形的數量代表物種於該樣本的族群規模。雖然紅方塊和綠圈圈的群集組成不同,但兩者的 unweighted UniFrac 為零。但細查後發現,紅方塊在系統發生樹左側的比例較高,而綠圈圈在右側的比例較。加總各枝對 β 多樣性的貢獻得出兩者的 weighted UniFrac 為 0.11。
UniFrac distance 有什麼前提與含義?
UniFrac 的前提是 16S rRNA 序列相似度與微生物的生態棲位相關
處於相異環境的族群會受不同的選擇壓力而逐漸分歧。環境差異愈大,族群間隔閡愈深,累積的遺傳差異也愈多。依此假設,兩族群在系統發生樹上的距離,反映了它們的生態棲位差距。
在 UniFrac 這類歧異度指標當中 (divergence-based metric),群集差異由其中的族群相異程度而定。群集成員的生態棲位差異愈大,則群集間的 β 多樣性也愈高。基於用 16S rRNA 重建的系統發生樹,UniFrac 的數值意味著不同微生物群集的適應性特徵差異。
然而,UniFrac 的前提不盡合理
然而,16S rRNA 之所以被選為分類鑑定的基因,正是因為它是核醣體的部件,攸關個體存續,所以在演化中的改變幅度較小。因此,16S rRNA 應該與微生物在特定環境的適存度較無關聯。
其次,頻繁的水平基因移轉使得微生物除了不可或缺的核心基因體 (core genome) 外,還附帶功能多樣的基因體 (accessory genome)。這些附帶基因體的組成多變且易受環境影響,所以即使某物種具有相同的核心基因體,也可能因為具有不同的附帶基因體,而表現出迥然不同的特性。
鑒於核心基因 的穩定性和水平基因移轉的多變性,以 16S rRNA 建立的系統發生樹能否忠實反映微生物的生態棲位便很可議了。
此外,UniFrac 的衍伸意義模糊
因此 UniFrac 其實只反映了群集在 16S rRNA 以及與其密切相關之基因的差異,無法將解釋推廣到包含水平基因移轉在內的其餘基因體。儘管 Lozupone & Rob Knight (2008) (UniFrac 開發者)主張,UniFrac 提供的資訊仍多過只基於分類而不考慮序列相似度的指標。
但究竟有多少基因與 16S rRNA 連鎖,使用者的推論能衍伸到什麼程度,也不是單從 UniFrac 可以得知。因此,即使 UniFrac 是量化的 β 多樣性指標,其延伸含義的解釋空間仍然很模糊。
使用 UniFrac 對分析結果有何影響?
不同的多樣性指標賦予序列不同權重
若不論生態學意義,其實各項多樣性指標都用了不同手段來壓低偽序列的影響。以採用定量資料的指標為例,由於樣本內的稀有序列往往是建庫或定序時引入的偽序列,所以這些指標賦予稀有序列較低的權重,以免偽序列誇大多樣性的估計值。
除了數量,核酸序列亦是判斷偽序列的依據。在品質較好的資料裡,偽序列往往與正確序列只有幾個鹼基差異。然而,若使用 Jaccard distance 或 Bray-Curtis dissmilarity 等以分類為基礎的 β 多樣性指標,會把微量差異視為獨立序列,以致於深受資料品質影響。
UniFrac 能緩衝技術誤差的影響
相較之下,UniFrac distance 採計序列的差異。相似序列對 β 多樣性的貢獻少,歧異序列對 β 多樣性的貢獻大,所以較能容忍定序過程中引進的誤判,或是雙端定序合併時造成的長度不一。因此,當研究迥異的環境微生物,或是僅有低品質的資料時,UniFrac 能緩衝干擾與技術誤差的影響。
UniFrac distance 會壓低組間差異
不過,前述特性也降低了 UniFrac 的靈敏度。當 (1) 環境差異不大,選擇壓力沒有體現在微生物的基因體,或是 (2) 資料品質優良,建庫和定序誤差的衝擊微弱時,UniFrac 這樣保守的策略會降低發現組間差異的機會。
UniFrac 的優缺點是什麼?
綜上所述,UniFrac 的優點是能充分利用標的基因的資訊,推論群集間各族群的生態棲位差異。此外,由於UniFrac 的數值依序列相似度加權,所以能緩衝技術誤差的影響。然而 UniFrac 的衍伸含義模糊,而且靈敏度較低,所以 Edgar 建議以直觀的 Bray-Curtis dissimilarity 取代 UniFrac。
可是 Bray-Curtis dissimilarity 雖然容易理解,它卻屬於非度量指標 (non-metric),也深受採樣不均影響。是以,各項指標皆有其優劣和適用條件,不同的指標能反映群集的不同特性,得協助我們了解研究對象的全貌。
何況實際研究時,往往無法在事前得知哪些方法有效,而是測試不同指標後才找到值得關注的現象。所以我認為,在欠缺研究脈絡的情況下執著於特定指標,是放棄發現新關聯的機會。
比起選擇方法,我相信更重要的是詮釋(如前所述,方法沒得挑,總是有什麼都拿來試試看)。其實在 Edgar 的文章也不斷強調,要區分指標數值和其衍伸含義。在標的基因分析裡,各項指標依據的不是微生物群、不是細菌、甚至不是核酸本身,而是定序後獲得的鹼基序列。
無論是物種觀念、親緣關係還是代謝功能分析,都是基於理論由序列推論出生物含義。隨著嵌套的假設增加,指標含義也愈趨複雜,以至於結果難以詮釋、容易誤解或不易檢驗。
因此,即便想使用 UniFrac 這類涵蓋系統發生資訊的指標,也要從最直觀的指標著手,才有衡量複雜指標的基線。掌握最底層的序列含義後,才不至於把推論建立在虛妄的根基。
參考資料
- Betadiversity OTU picking vs. non OTU picking (DADA2). Qiime2 forum. (Accessed Feb 5, 2020)
- C. Lozupone and R. Knight. (2005) UniFrac: a New Phylogenetic Method for Comparing Microbial Communities. Appl Environ Microbiol.
- C. Lozupone and R. Knight. (2008) Species Divergence and the Measurement of Microbial Diversity. FEMS Microbiol Rev.
- J. Prosser et al. (2007) The role of ecological theory in microbial ecology. Nat Rev Microbiol.
- Why use UniFrac? Uearch. (Accessed Feb 2, 2020)
- Does it make sense to use weighted unifrac distances for 16s data? Qiime2 forum. (Accessed Feb 2, 2020)
補充
本文對於 UniFrac 的解釋和理論背景參考指標發明者的論文:
- C. Lozupone and R. Knight. (2005) UniFrac: a New Phylogenetic Method for Comparing Microbial Communities. Appl Environ Microbiol.
- C. Lozupone and R. Knight. (2008) Species Divergence and the Measurement of Microbial Diversity. FEMS Microbiol Rev.
本文關於 UniFrac 前提有疑慮且靈敏度不足的解釋延伸自 Edgar 的評論:
Why use UniFrac? (https://drive5.com/usearch/manual/unifrac_comments.html)
核心基因體 (core genome) 和附帶基因體 (accessory genome) 之別:
J. Prosser et al. (2007) The role of ecological theory in microbial ecology. Nat Rev Microbiol.
至於為什麼 UniFrac 屬於 phylogenetic analysis 可參考:
- Does it make sense to use weighted unifrac distances for 16s data? (https://forum.qiime2.org/t/does-it-make-sense-to-use-weighted-unifrac-distances-for-16s-data/12768/2)
- Betadiversity OTU picking vs. non OTU picking (DADA2) (https://forum.qiime2.org/t/does-it-make-sense-to-use-weighted-unifrac-distances-for-16s-data/12768/2)